Анализ - ближайший сосед
Cтраница 1
Анализ ближайших соседей - методический прием, при помощи которого удается определите последовательность нуклеотидов на определенном участке нуклеиновых кислот. Вначале создают систему, содержащую РНК-полимеразу, ДНК-матрицу и все четыре нуклеозидпирофосфата, один из которых, например АТФ, меченый по внутреннему фосфату. Синтезированную РНК затем гидролизуют щелочью, в результате чего радиофосфор оказывается связанным с соседним нуклеозидом. Этот нуклео-зид может быть любым из четырех возможных. Далее разделяют образовавшиеся рибонуклеозидмонофосфаты, определяют их радиоактивность и по ней судят о частоте, с которой адениловый остаток следует за данным соседом в цепи РНК. Потом опыт повторяют трижды, каждый раз вводя в систему новый меченый нуклеозидтрифосфат, вследствие чего получают частоты всех шестнадцати возможных пар, сконструированных из ближайших соседей. Найденные таким образом величины весьма хорошо согласуются с соответствующими данными для ДНК-матрицы. [1]
 |
Этапы созревания рРНК. [2] |
Анализ ближайших соседей в ДНК различного происхождения свидетельствует в пользу противоположной полярности. [3]
Что говорит нам анализ ближайшего соседа в отличие от анализа квадратов. [4]
Определение соотношения оснований в полученной РНК путем анализа ближайшего соседа ( меченные Р32 субстраты) при использовании ДНК-затравки с резко отличающимся нуклеотидным составом показало, что образующаяся РНК комплементарна затравочной ДНК - При использовании олиготимидиловой кислоты в качестве затравки из смеси рибонуклеозид-5 - трифосфатов в реакции участвует лишь аденозин-5 - трифосфат с образованием полиадениловой кислоты. Щелочной гидролиз ( сопровождающийся переносом Р32) показал, что адениловые и урациловые остатки противостоят друг другу, а отсюда затравка определяет как состав, так и последовательность оснований в продукте. Для частично очищенной бактериальной РНК-полимеразы затравкой могут служить полирибонуклеотиды; возможно, при этом функционирует не ДНК-зависимая полимераза, а какой-то другой фермент. [5]
Существует два метода анализа, результаты которых указывают на последовательность оснований в молекулах ДНК - Однако сами по себе они не обеспечивают полного определения нуклеотидной последовательности в ДНК. Один из этих методов - анализ ближайших соседей, а другой - анализ пиримиди-новых блоков ДНК. Применяя второй метод, ДНК обрабатывают разбавленной неорганической кислотой. В этих условиях отщепляются пуриновые основания а на их местах в полимере остаются дезоксирибозофосфатные звенья. Что же касается пиримндиновых нуклеотидов, то они остаются на исходных местах в молекуле ДНК. [6]
Из всех этих данных вытекает, что затравочная ДНК определяет не только суммарный состав образующейся РНК, но и последовательность оснований в ней. Этот вывод подтверждается также результатами анализа ближайших соседей [21, 22], о котором мы уже писали ( стр. [7]
Распределение пар линий может быть определено во многом подобно тому, как мы поступали с точками, хотя вычисления несколько усложняются, так как, в отличие отточек, линии имеют размерность. Может показаться, что следует просто выбрать центр каждой линии и провести анализ ближайшего соседа для этих точек. Однако, вследствие того, что линии имеют различные длины, эта процедура не даст нам правдивой картины распределения самих линий. С точки зрения статистики часто считается полезным делать случайную выборку. Затем мы измеряем эти расстояния и подсчитываем среднее РБС. [8]
 |
Расстояние до ближайшего соседа среди линий. Поиск ближайшего соседа между линиями с использованием случайно выбранной точки на одной из них. [9] |
Кроме того, чтобы критерий был полезен, линии должны быть по меньшей мере в полтора раза длиннее среднего расстояния между ними. Если количество линий в покрытии мало, оценка плотности, используемая в анализе ближайшего соседа должна быть скорректирована весовым коэффициентом ( п - 1) / п, где п - число линий распределения. [10]
 |
Расстояние до ближайшего соседа среди линий. Поиск ближайшего соседа между линиями с использованием случайно выбранной точки на одной из них. [11] |
Кроме того, чтобы критерий был полезен, линии должны быть по меньшей мере в полтора раза длиннее среднего расстояния между ними. Если количество линий в покрытии мало, оценка плотности, используемая в анализе ближайшего соседа должна быть скорректирована весовым коэффициентом ( п - 1) / п, где п - число линий распределения. [12]
Для полного установления строения нуклеиновых кислот необходимо, помимо установления основного принципа построения полимера, выяснения структуры входящих в него компонентов и определения их количества в составе полимера, решить вопрос о последовательности мономерных единиц в полимерной цепи. Современные данные показывают, что полинуклеотидная цепь нуклеиновых кислот, за исключением ДНК-спутника из крабов [ в основном поли - ( 1 ( Ар Тр) ] и, возможно, некоторых других ДНК-спутников, не построена из повторяющихся звеньев. С другой стороны, как показывают результаты анализа ближайших соседей, распределение нуклеотидных единиц в цепи сильно отличается от среднестатистического. В специфической последовательности нуклеотидов в цепи ДНК содержится, по современным биохимическим представлениям, вся информация о последовательности аминокислот в белках данного организма. [13]
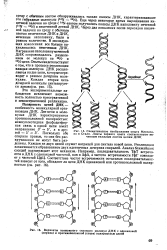
Поскольку обе спирали правые, то они без раскручивания не могут быть разделены. Каждая из двух цепей служит матрицей для синтеза новой цепи. Репликация заканчивается образованием двух идентичных дочерних спиралей. Анализ ближайших соседей подтверждает этот механизм. [15]
Страницы: 1 2